Nature Microbiology | 大型语言模型改进原核病毒蛋白质注释
引言:微生物世界的多样性和功能一直是科学家们关注的焦点,然而,宏基因组样本中病毒基因组的注释一直是一个挑战。现有的方法受到特征病毒蛋白质缺乏和病毒序列差异的限制,这妨碍了对病毒多样性和功能的深入理解。本研究证明了蛋白质语言模型(PLMs)在改善病毒蛋白质注释方面的潜力,为我们提供了揭示微生物世界新视角的工具。观点:蛋白质语言模型:捕获原核病毒蛋白质功能的新途径功能预测:拓展病毒蛋白质注释的空间数据
引言:微生物世界的多样性和功能一直是科学家们关注的焦点,然而,宏基因组样本中病毒基因组的注释一直是一个挑战。现有的方法受到特征病毒蛋白质缺乏和病毒序列差异的限制,这妨碍了对病毒多样性和功能的深入理解。本研究证明了蛋白质语言模型(PLMs)在改善病毒蛋白质注释方面的潜力,为我们提供了揭示微生物世界新视角的工具。
观点:
蛋白质语言模型:捕获原核病毒蛋白质功能的新途径
功能预测:拓展病毒蛋白质注释的空间
数据分析:解读病毒多样性与功能的关系
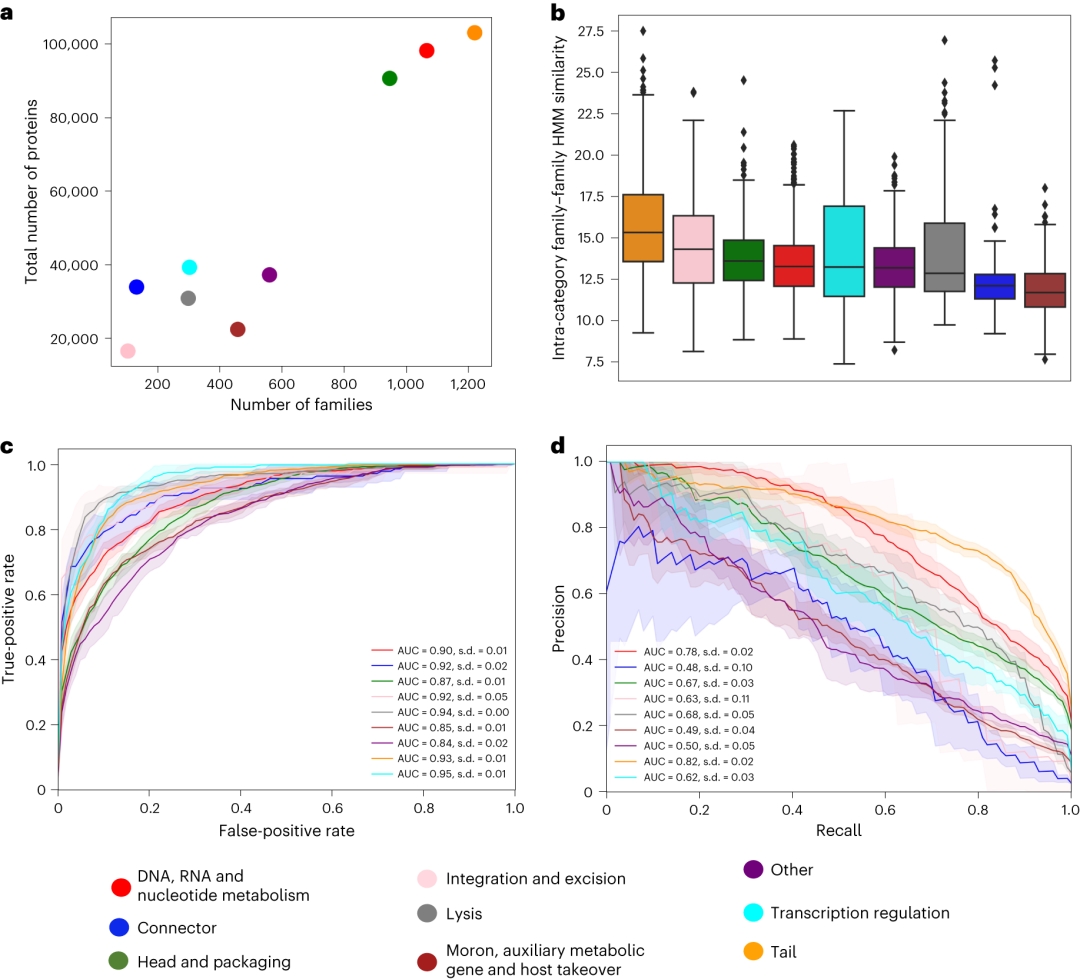
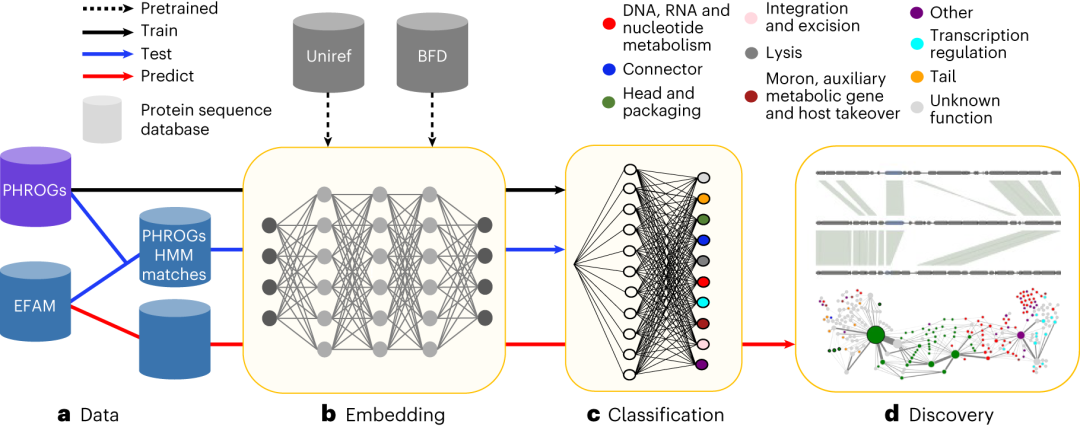
正文:在本研究中,我们利用蛋白质语言模型(PLMs)的强大功能,成功改进了原核病毒蛋白质的注释。通过对全球海洋病毒组数据的分析,我们发现,使用PLMs进行功能预测可以将病毒蛋白质家族的注释部分扩展了29%,为我们提供了更为全面的理解。特别是,我们成功地识别出了一些以前未注释的序列中的重要功能,例如海洋超微蓝藻中的移动元件的整合酶和全球广泛传播的病毒元件的衣壳蛋白。通过改进的高级功能注释,我们不仅拓展了病毒蛋白质的远程同源性检测,还为研究人员提供了一种表征不同病毒序列之间基因组组织相似性的方法。
我们进一步分析了基于PLMs的蛋白质嵌入对病毒蛋白质功能分类的影响。我们的结果显示,PLMs可以有效地将病毒蛋白质分为不同的功能类别,并且与传统方法相比,具有更高的准确性和灵敏度。此外,我们利用PLMs的功能分类器验证了在海洋病毒组中挑选的病毒蛋白质的功能,进一步证明了PLMs在改进病毒蛋白质注释方面的有效性。
结语:本研究通过利用蛋白质语言模型(PLMs)的先进功能,成功改进了原核病毒蛋白质的注释。我们的研究揭示了一种全新的解析微生物世界的方法,为研究人员提供了更为全面和准确的工具。这一发现不仅对于理解微生物多样性和功能具有重要意义,还为生物信息学领域的发展提供了新的思路和方法。
Flamholz, Z.N., Biller, S.J. & Kelly, L. Large language models improve annotation of prokaryotic viral proteins. Nat Microbiol (2024). https://doi.org/10.1038/s41564-023-01584-8
宏基因组推荐
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
猜你喜欢
iMeta高引文章 fastp 复杂热图 ggtree 绘图imageGP 网络iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文

魔乐社区(Modelers.cn) 是一个中立、公益的人工智能社区,提供人工智能工具、模型、数据的托管、展示与应用协同服务,为人工智能开发及爱好者搭建开放的学习交流平台。社区通过理事会方式运作,由全产业链共同建设、共同运营、共同享有,推动国产AI生态繁荣发展。
更多推荐
 已为社区贡献7条内容
已为社区贡献7条内容







所有评论(0)