DNA语言模型,100张卡能做,1张卡也能做?
最近,大语言模型在生物领域进展迅速。在DNA,RNA,蛋白质领域都有各种各样的大模型推出。相信各位从事AI+生物医学研究的小伙伴,对这些强大的模型也十分眼馋。不过,由于大部分研究组的计算资源都比较有限,因此许多小伙伴可能会“望大模型兴叹”。不过,今天要讲述的故事,将告诉大家,虽然许多大模型是由坐拥成千上万张GPU的大团队完成,拥有相对较少资源的团队,也有机会训练自己的大模型,并且发表在顶级的杂志上

最近,大语言模型在生物领域进展迅速。在DNA,RNA,蛋白质领域都有各种各样的大模型推出。相信各位从事AI+生物医学研究的小伙伴,对这些强大的模型也十分眼馋。不过,由于大部分研究组的计算资源都比较有限,因此许多小伙伴可能会“望大模型兴叹”。
不过,今天要讲述的故事,将告诉大家,虽然许多大模型是由坐拥成千上万张GPU的大团队完成,拥有相对较少资源的团队,也有机会训练自己的大模型,并且发表在顶级的杂志上!
过去两个月内,两项与DNA语言大模型相关的研究相继在顶尖学术杂志上发表。2024年11月15日,Arc研究所的Patrick D. Hsu和Brian L. Hie团队在国际顶尖期刊《Science》上发表了题为《Sequence modeling and design from molecular to genome scale with Evo》的研究论文,该论文被选为当期的封面文章。(新物种、新 CRISPR 系统!Evo 大模型突破全基因组生成,创造生物大模型新标杆)这项研究展示了Evo模型在解码和设计从分子到基因组尺度的对象方面的无与伦比的准确性,可能彻底改变合成生物学的工作方式。
与此同时,2024年10月30日,Broad Institute的Shao Bin博士与独立研究者Yan Jiawei博士在《Nature Communications》杂志上发表了题为《A long-context language model for deciphering and generating bacteriophage genomes》的最新研究成果,推出了生成式DNA大语言模型megaDNA。该模型利用无标注的噬菌体基因组数据进行预训练,不仅能够准确预测噬菌体的必需基因,还能生成长达10万碱基对的新基因组片段,展示了如同自然语言写作般生成DNA序列的能力。此外,模型在学习过程中获得的深层信息(embedding)还可应用于蛋白质突变效果预测、基因调控研究以及无标注DNA片段分类等多个下游任务,展现出良好的泛化能力。尽管这两项DNA语言模型研究均发表在顶级期刊上,但两者的团队规模和计算资源却大相径庭。本期我们将从模型参数、计算资源和模型功能等多个角度分析DNA大语言模型的研究现状、机遇与挑战。
Evo: 利用上百张GPU炼成强大工具!
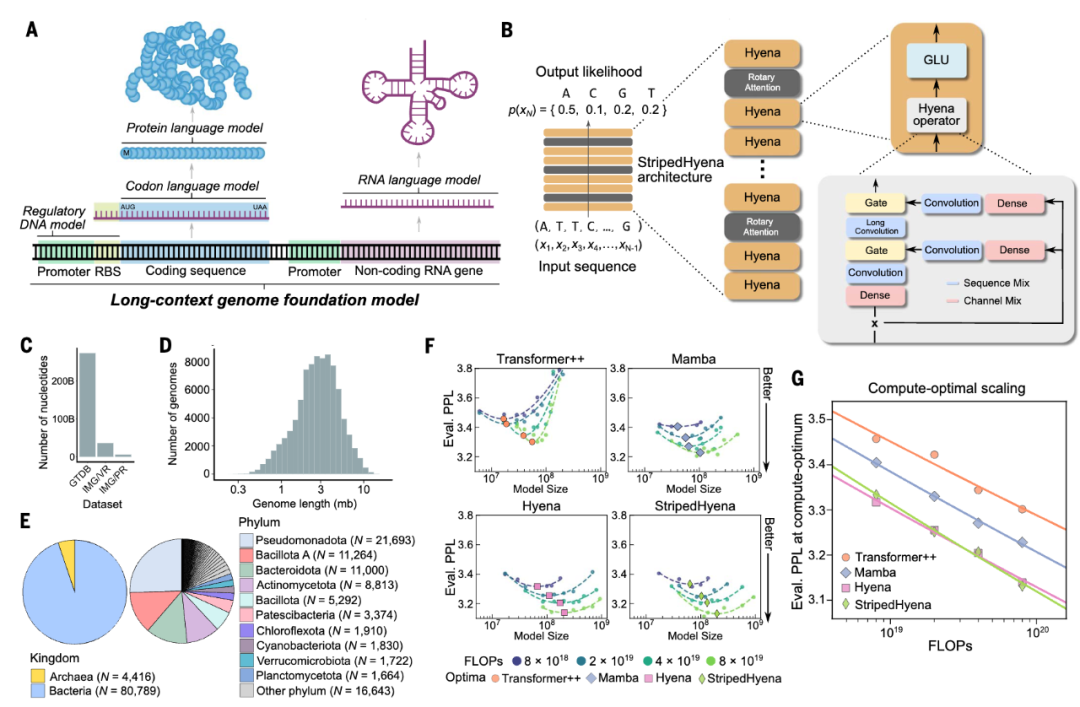
Evo使用了StripedHyena架构[1],在270万个进化多样的原核生物和噬菌体基因组上进行了预训练,涵盖了3000亿个核苷酸token,从而获得对遗传语言的基本理解。Evo能够生成超过1兆碱基长度的具有合理基因组结构的DNA序列。此外,Evo的参数规模达到70亿,最大上下文长度可达131072个token,这使其能够揭示编码序列与非编码序列之间错综复杂的共同进化关系,设计出复杂的生物系统,如CRISPR-Cas复合物和IS200与IS605转座子。
如上图B所示,StripedHyena架构结合了注意力机制和数据控制的卷积算子,能够有效处理和学习长序列中的模式。同时,该模型将上下文窗口扩展至长达130000碱基,显著提高了模型识别基因与其他基因调控元件(如启动子和增强子等)之间关系的能力。为了确定Evo的最佳架构和缩放比例,上图F和G比较了在计算最优边界上预训练的不同模型的缩放率,旨在实现数据集大小与模型大小之间的最佳计算分配。为防止恶意用户设计生物武器,研究人员从AI的训练集中删除了任何可能攻击人类或其他真核生物的病毒序列,并使用100块A100 GPU在接近3000亿核苷酸序列信息上进行了为期4周的训练。
总而言之,Evo是一个利用大量GPU炼成的强大的基因组建模工具,拥有预测、生成和设计整个基因组序列的能力,不仅为生命科学提供了新的理论支撑,还有望在基因编辑、药物发现、疾病诊断和农业等领域发挥重要作用,助力多个领域的突破性成果研发。
megaDNA: 只有一张卡,也能炼成小而美的领域工具!
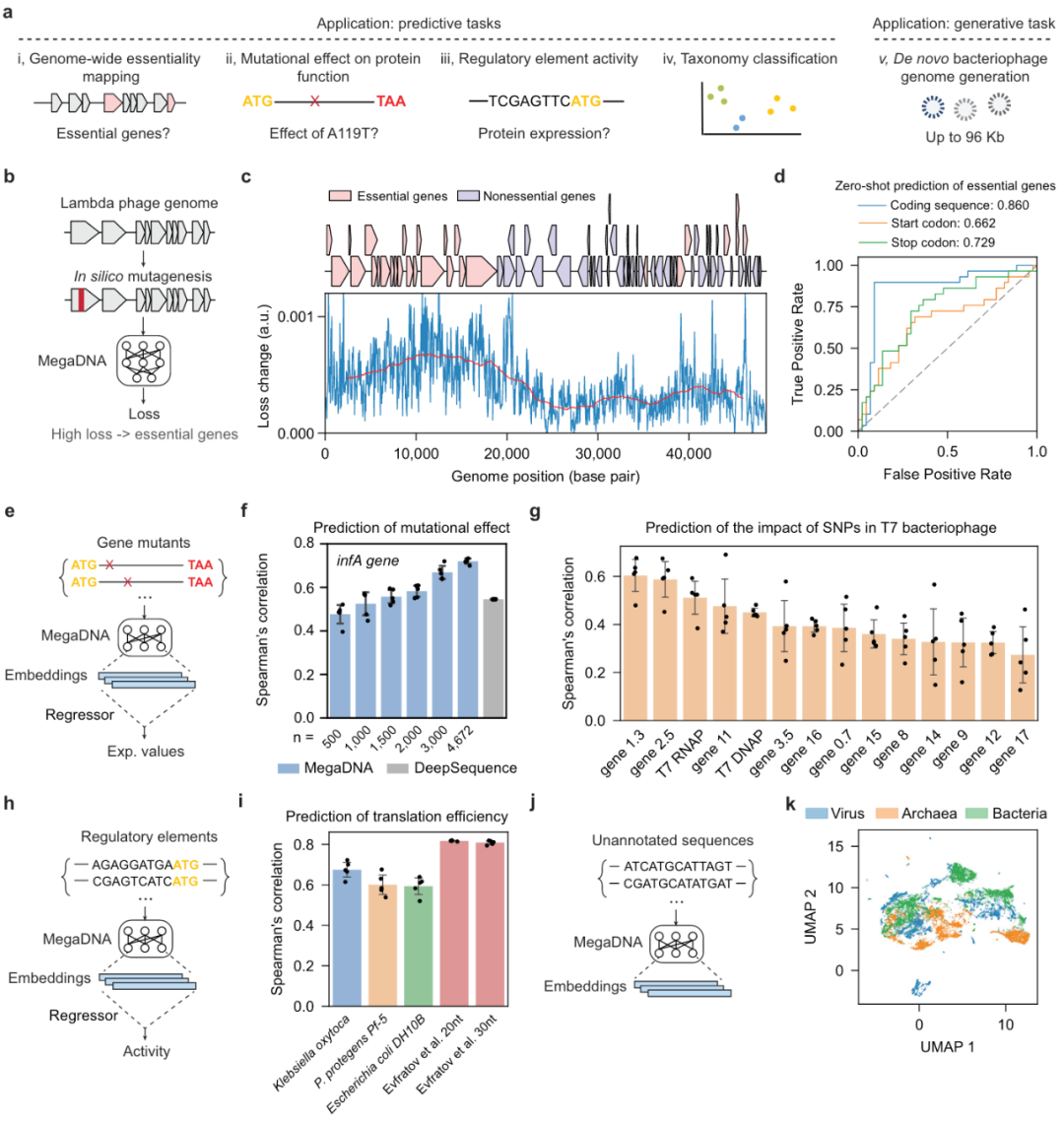
与Evo相比,megaDNA采用了“小而精”的策略, 聚焦于噬菌体的基因组,利用NCBI GenBank等公开数据库中的约10万个无标注高质量噬菌体完整基因组作为训练数据。类似于Evo,megaDNA在分词环节并未使用传统的 BPE分词器或将DNA切分为k-mer,而是将每个碱基视为独立的token,从而避免了分词过程中可能带来的偏差,并使语言模型能够学习到单碱基精度的DNA调控和设计信息。
在模型架构上,megaDNA借鉴了Meta公司开发的多层Transformer架构[2],包含1.5亿个参数。每层的注意力机制用于处理不同精度的DNA信息,从而克服了传统Transformer模型只能处理短序列的限制。megaDNA能一次性分析长达96000个碱基的DNA序列,足以覆盖大多数噬菌体的完整基因组。
作者利用megaDNA模型生成了1000条崭新的基因组DNA序列,并通过软件geNomad对这些序列进行了系统评估。这些人工生成的基因组序列平均长度为4.3万碱基对,包含67个预测基因,这些特征与训练数据中的真实噬菌体基因组高度相似。其中,22%的序列被识别为有尾噬菌体(Caudoviricetes)。
在表达调控方面,这些序列具备完整的潜在功能基因表达系统,包括典型的细菌启动子序列(-35 和 -10 区),其5'端非翻译序列的转录活性显著高于随机DNA。同时,在起始密码子ATG前,具有富含A和G碱基的核糖体结合位点(RBS)。通过EMSFold进行结构预测,结果显示这些预测的基因能够形成有效的蛋白质折叠结构。在功能方面,预测基因编码了噬菌体所需的关键功能蛋白,涉及尾部结构、DNA代谢、头部组装和细胞裂解等功能。值得一提的是,这些生成的基因组序列及其预测基因与已有的训练数据具有极低的序列相似性。
对比
机遇与挑战
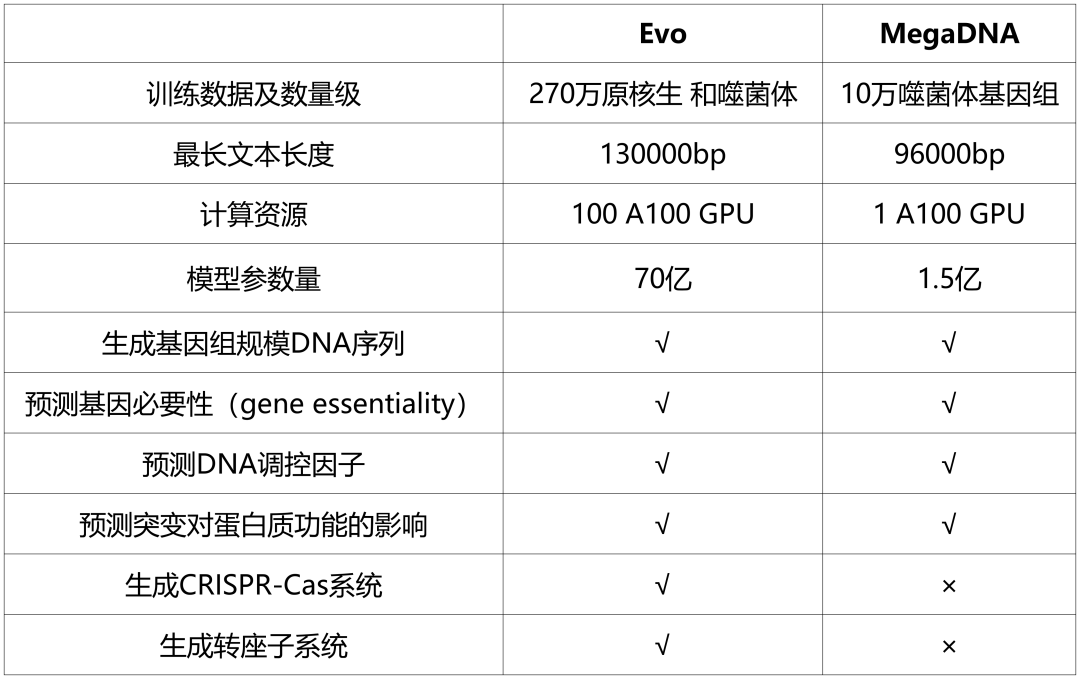
Evo和megaDNA代表了两种不同的研究风格。从共同点来看,Evo和megaDNA都可以帮助理解噬菌体基因组,挖掘其中基因的具体功能,包括突变如何影响蛋白质功能。Evo模型的参数量更大,训练数据也更丰富,涵盖了噬菌体基因组,能够生成CRISPR-Cas系统和转座子系统。如果进一步在实验上验证和优化这些生成的系统,可能为基因编辑提供新的工具。而megaDNA则专注于领域内的高质量数据,尽管数据量有限,却取得了不错的效果。这说明不止是坐拥大量资源的研究组才能训练大模型, 对于资源有限的研究组,如果能定义好感兴趣的研究问题,收集高质量的数据,即使是训练小模型,也能取得不错的效果。
对于噬菌体基因组这一特定问题而言,虽然megaDNA和Evo都能够处理数量级达到10万碱基对的上下文,但原核生物的基因组一般在百万碱基对水平, 例如大肠杆菌MG1655菌株的基因组为460万碱基对,这相差了一个数量级。因此,若读长不足以覆盖整个细菌基因组,可能会导致一些问题。对于真核生物而言,情况更加复杂。真核生物基因组中约99%是非编码序列,包含大量“垃圾”序列,因此使用语言模型生成真核基因组仍面临挑战。在有限的计算资源下,探索能够覆盖更长序列的模型依然是一项严峻的任务。
引用
[1]M. Poli et al., StripedHyena: Moving Beyond Transformerswith Hybrid Signal Processing Models, GitHub (2023);
https://github.com/togethercomputer/stripedhyena.
[2]Yu, L. et al. Megabyte: Predicting million-byte sequences with multiscale transformers. Adv. Neural Inf. Process Syst. 36, 78808–78823 (2023).
高颜值免费 SCI 在线绘图(点击图片直达)
最全植物基因组数据库IMP (点击图片直达)
往期精品(点击图片直达文字对应教程)



魔乐社区(Modelers.cn) 是一个中立、公益的人工智能社区,提供人工智能工具、模型、数据的托管、展示与应用协同服务,为人工智能开发及爱好者搭建开放的学习交流平台。社区通过理事会方式运作,由全产业链共同建设、共同运营、共同享有,推动国产AI生态繁荣发展。
更多推荐
 已为社区贡献9条内容
已为社区贡献9条内容




































所有评论(0)