近红外数据nirs数据处理-1
近红外资料的收集,对于nirs数据的结构的分析,对于布局文件的修正。对homer2的处理流程函数的说明。
此处先给出学习资料的链接:
卢家峰课程的主页
B站搬运版
使用mne进行处理(b战搬运)
mne处理的youtube的链接
jupyter的代码链接(可能打不开)
近红外可以处理的方向
使用matlab的插件工具进行处理
手把手教你使用NIRS_KIT
Homer2
有homer3的版本,但是使用总是有问题。可能版本不太符合。
记录本人处理.nirs过程的一些过程:
数据导入
之前希望使用mne进行数据读取,但是现在的mne合并了mne_nirs工具,读取的是snirs后缀的文件,.snirs需要使用homer3(下载matlab插件进行自动转换)对.nirs的数据进行默认转换。是一种共享格式的近红外数据。
此外还有.hcx的后缀的,使用nirspark的软件可以处理,该软件并未开源。
可能按照homer2的处理逻辑来进行组织。
进行文件读取和数据解析。
Homer2这个给出的说明比较详尽了。
%通过这个命令,读取到工作区中
load(".nirs","-mat")
import scipy.io
import os
import numpy as np
data_dir=r"文件夹路径"
file_path = '文件名.nirs'
file_path=os.path.join(data_dir,file_path)
data = scipy.io.loadmat(file_path)
同样,我们也可以使用python来读取下面这个对象为字典结构。
可以参考matlab的结构体的形式来访各种数据。
数据说明
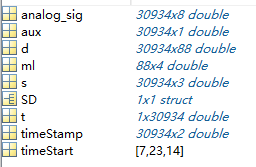
以这个读取的例子为例。
在MATLAB中导入NIRS(近红外光谱)数据后,工作区中的变量通常包含与近红外光谱测量相关的信息。以下是对这些变量的可能解释:
-
analog_sig (30934x8 double): 这可能是模拟信号数据,表示从近红外设备中采集的原始信号。矩阵的维度为30934行(时间点)和8列(可能是不同的通道或波长)。只有一行存在数据。意义不明,视为无效数据。
-
aux (30934x1 double): 这可能是辅助信号数据,通常用于记录额外的信息,如触发信号或其他同步数据。
-
d (30934x88 double): 这可能是处理后的光谱数据,表示在不同波长或通道下的光强度变化。矩阵的维度为30934行(时间点)和88列(可能是不同的波长或通道)。
-
ml (88x4 double): 这可能是测量列表(Measurement List),用于描述不同通道的配置信息,如光源、探测器和波长的组合。
-
s (30934x3 double): 这可能是系统状态或标记数据,用于记录实验过程中的事件或状态变化。
-
SD (1x1 struct): 这可能是源-探测器结构(Source-Detector Structure),包含关于光源和探测器的配置信息,如它们的位置和距离。
-
t (1x30934 double): 这可能是时间向量,表示每个数据点对应的时间戳。
-
timeStamp (30934x2 double): 这可能是时间戳数据,用于记录每个数据点的具体时间信息。
-
timeStart ([7,23,14]): 这可能是实验开始的时间,格式可能为[小时, 分钟, 秒]。
通道的说明
说明为什么是88个通道,有18个发射器和15个接收器。
下面两幅图应该是镜像的。
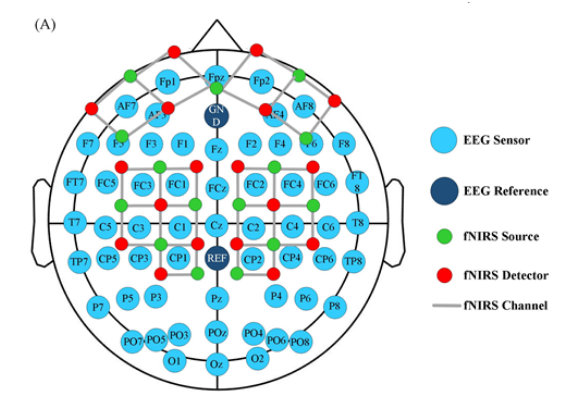
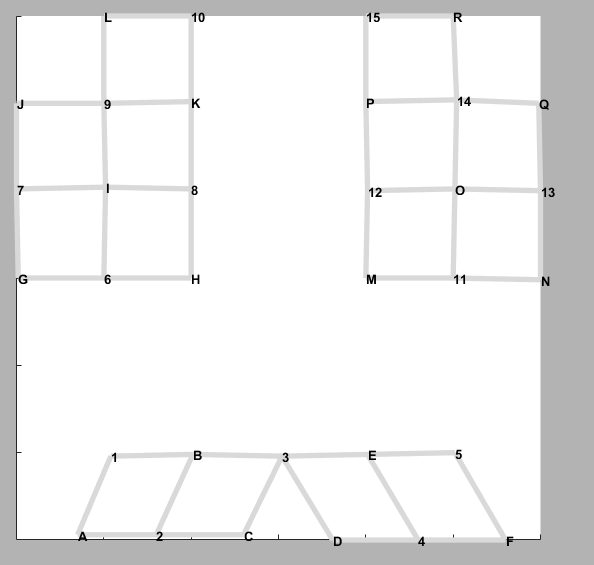
-
FC1: 位于额叶(Frontal, F)和中央叶(Central, C)之间,靠近中线(1 表示靠近中线)。这个区域与运动规划和执行相关。
-
FC3: 位于额叶和中央叶之间,靠近左侧(3 表示左侧)。这个区域也与运动功能相关,但更偏向于左侧大脑半球。
-
FC5: 位于额叶和中央叶之间,更靠近左侧(5 表示更外侧的位置)。这个区域可能涉及更具体的运动控制和感觉处理。
-
C1: 位于中央叶(Central, C),靠近中线(1 表示靠近中线)。这个区域与初级运动皮层和感觉处理相关。
-
C3: 位于中央叶,靠近左侧(3 表示左侧)。这个区域与左侧大脑半球的运动控制和感觉处理相关。
-
CP1: 位于中央叶(Central, C)和顶叶(Parietal, P)之间,靠近中线(1 表示靠近中线)。这个区域与感觉整合和运动协调相关。
AF1、FP1、FPz、FP2 和 AF8 是脑电图(EEG)电极放置中的位置,通常用于国际 10-20 系统或其扩展的 10-10 系统。
-
AF1:
- AF 表示“Anterior Frontal”,即前额叶区域。
- 1 表示靠近中线。
- AF1 位于前额叶,靠近中线,通常与高级认知功能、情绪调节和决策制定相关。
-
FP1:
- FP 表示“Frontopolar”,即额极区域。
- 1 表示靠近左侧。
- FP1 位于额极区域,靠近左侧,通常与额叶的前部活动相关,涉及高级认知功能和情绪处理。
-
FPz:
- FP 表示“Frontopolar”,即额极区域。
- z 表示中线位置。
- FPz 位于额极区域的正中线,通常用于参考电极或研究中线额叶活动。
-
FP2:
- FP 表示“Frontopolar”,即额极区域。
- 2 表示靠近右侧。
- FP2 位于额极区域,靠近右侧,通常与右侧额叶的前部活动相关,涉及高级认知功能和情绪处理。
-
AF8:
- AF 表示“Anterior Frontal”,即前额叶区域。
- 8 表示靠近右侧。
- AF8 位于前额叶,靠近右侧,通常与右侧前额叶的活动相关,涉及高级认知功能和情绪调节。
这些电极位置通常用于研究大脑前额叶区域的活动,特别是在认知、情绪和决策任务中。具体的功能可能会根据具体的研究任务和个体差异有所不同。
其中9所在的区域应该是额叶和中央叶的部分。一般涉及运动和感觉协调的部分。14是另一个半脑的对应区域。
前侧是进行决策认知的区域。
因为发射器发射两种频谱的光线。所以(15+15+14)* 2 记录了88个通道的数据。
其中和d对应存储的数据的描述在ml文件中存储。
第三列不是非常明确意义,可能是权重信息或什么,因为都是1。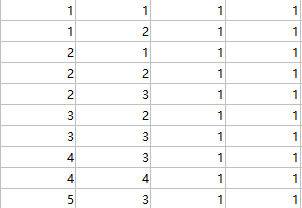
采样率的确定
读取数据需要采样率,结构体中没有FS数据 ,但是有t这个时间记录。
我们可以知道是11HZ。
根据homer2的处理逻辑,在开始是不会有绘图的。因为需要通过某个处理过程,对数据进行操作的计算,不然是没有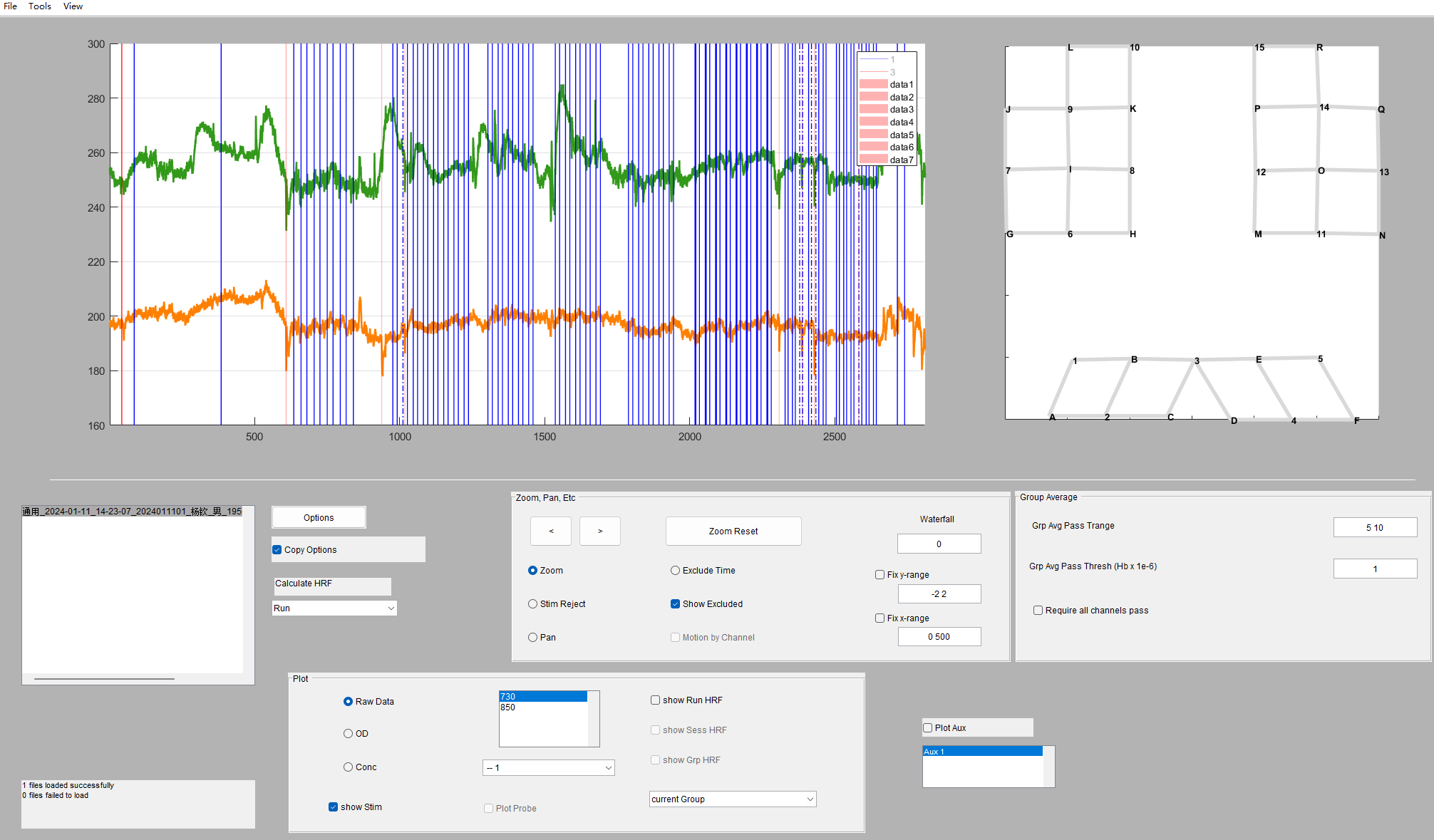
经过处理的数据有了procResult和procInput,以及tIncMan和userdata等额外的数据。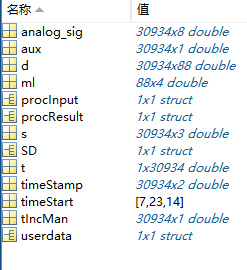
我们可以通过这个过程来对数据进行处理。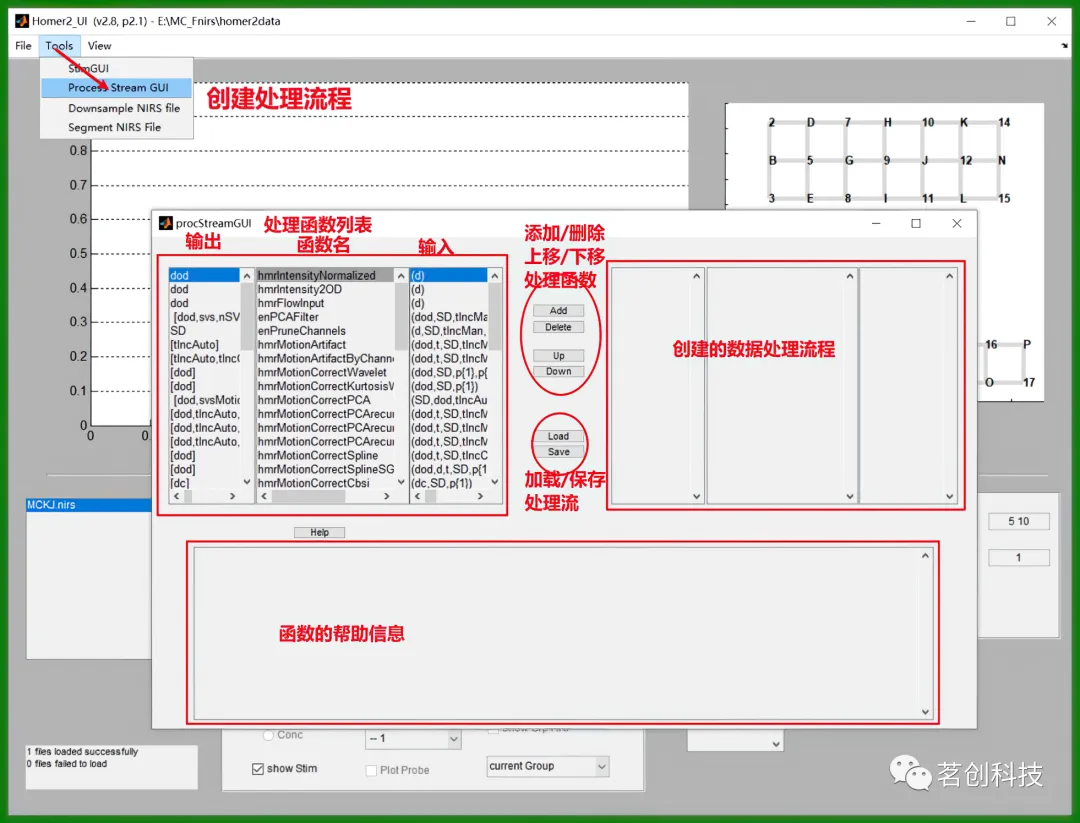
我们可以看到这些函数的参数,函数名和输出结果。从而查看和修改这些函数。
先对这些可能涉及的参数进行说明:
在近红外光谱(NIRS)数据处理中,这些参数通常用于描述光强度、光密度、血红蛋白浓度、信号质量等信息。以下是这些参数的详细说明:
1. d
- 含义:原始光强度数据。
- 说明:
d是一个矩阵,通常大小为时间点数 × 通道数。- 每一列代表一个通道(即一个光源-探测器对)的光强度数据。
- 单位通常是任意单位(a.u.),表示探测器接收到的光强度。
2. dod
- 含义:光密度变化(ΔOD,Delta Optical Density)。
- 说明:
dod是通过对原始光强度数据d进行转换得到的。- 计算公式:
Δ O D = − log 10 ( I I 0 ) \Delta OD = -\log_{10}\left(\frac{I}{I_0}\right) ΔOD=−log10(I0I)
其中,( I ) 是当前光强度,( I_0 ) 是基线光强度(通常取实验开始时的平均值)。 dod是一个矩阵,大小与d相同,表示每个通道的光密度变化。
3. SD
- 含义:光源-探测器结构信息(Source-Detector Structure)。
- 说明:
SD是一个结构体,包含光源和探测器的位置信息以及通道的配置信息。- 主要字段:
SD.SrcPos:光源的位置坐标(单位:毫米,mm)。SD.DetPos:探测器的位置坐标(单位:毫米,mm)。SD.MeasList:通道列表,描述每个通道的光源和探测器索引。
SD用于描述实验的几何布局,是数据处理中的重要参数。
4. tIncMan
- 含义:手动标记的时间段(Time Inclusion Manual)。
- 说明:
tIncMan是一个逻辑向量,表示哪些时间段是有效的(1)或无效的(0)。- 通常用于手动标记需要排除的时间段(如运动伪影、设备故障等)。
- 大小与时间点数相同。
5. t
- 含义:时间向量。
- 说明:
t是一个列向量,表示每个时间点的时间戳(单位:秒,s)。- 用于描述数据的时间轴。
6. tIncChAuto
- 含义:自动标记的通道时间段(Time Inclusion Channel Auto)。
- 说明:
tIncChAuto是一个逻辑矩阵,表示每个通道在哪些时间段是有效的(1)或无效的(0)。- 通常通过自动算法(如信号质量检测)生成。
- 大小为
时间点数 × 通道数。
7. tIncAutoAux
- 含义:自动标记的辅助时间段(Time Inclusion Auto Auxiliary)。
- 说明:
tIncAutoAux是一个逻辑向量,表示哪些时间段是有效的(1)或无效的(0)。- 通常基于辅助信号(如加速度计、心率等)自动生成。
- 大小与时间点数相同。
8. dc
- 含义:血红蛋白浓度变化(Delta Concentration)。
- 说明:
dc是通过对dod数据进行计算得到的,表示氧合血红蛋白(HbO)和脱氧血红蛋白(HbR)的浓度变化。- 计算公式基于修正的比尔-朗伯定律(Modified Beer-Lambert Law):
Δ C = Δ O D d ⋅ ϵ \Delta C = \frac{\Delta OD}{d \cdot \epsilon} ΔC=d⋅ϵΔOD
其中, d d d 是光路径长度, ϵ \epsilon ϵ 是消光系数。 dc是一个三维矩阵,大小为时间点数 × 通道数 × 2,第三维分别表示 HbO 和 HbR。
9. s
- 含义:刺激标记(Stimulus Markers)。
- 说明:
s是一个矩阵,表示刺激事件的时间点和类型。- 通常大小为
时间点数 × 刺激类型数,每个元素表示某个时间点是否发生了某种刺激。 - 用于事件相关分析(如事件相关电位或事件相关血红蛋白响应)。
总结
| 参数 | 含义 | 大小/格式 |
|---|---|---|
d |
原始光强度数据 | 时间点数 × 通道数 |
dod |
光密度变化(ΔOD) | 时间点数 × 通道数 |
SD |
光源-探测器结构信息 | 结构体 |
tIncMan |
手动标记的时间段 | 时间点数 × 1 |
t |
时间向量 | 时间点数 × 1 |
tIncChAuto |
自动标记的通道时间段 | 时间点数 × 通道数 |
tIncAutoAux |
自动标记的辅助时间段 | 时间点数 × 1 |
dc |
血红蛋白浓度变化 | 时间点数 × 通道数 × 2 |
s |
刺激标记 | 时间点数 × 刺激类型数 |
其中p{}这些参数是函数额外的需求的参数,根据函数的功能是不同的。
SD布局文件的处理
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
# 提取发射源和检测器位置
SrcPos = data['SD']["SrcPos"][0][0] # 发射源位置
DetPos = data['SD']["DetPos"][0][0] # 检测器位置
# 创建 3D 图形
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
# 绘制发射源(红色)
ax.scatter(SrcPos[:, 0], SrcPos[:, 1], SrcPos[:, 2], c='r', marker='o', s=100, label='Sources')
for i, (x, y, z) in enumerate(SrcPos):
ax.text(x, y, z, f'S{i+1}', color='red', fontsize=12)
# 绘制检测器(蓝色)
ax.scatter(DetPos[:, 0], DetPos[:, 1], DetPos[:, 2], c='b', marker='s', s=100, label='Detectors')
for i, (x, y, z) in enumerate(DetPos):
ax.text(x, y, z, f'D{i+1}', color='blue', fontsize=12)
# 设置图形属性
ax.set_xlabel('X (cm)')
ax.set_ylabel('Y (cm)')
ax.set_zlabel('Z (cm)')
ax.set_title('Source and Detector Layout')
ax.legend()
# 显示图形
plt.show()
对默认的SD进行输出查看,因为布局的位置,会影响函数的处理。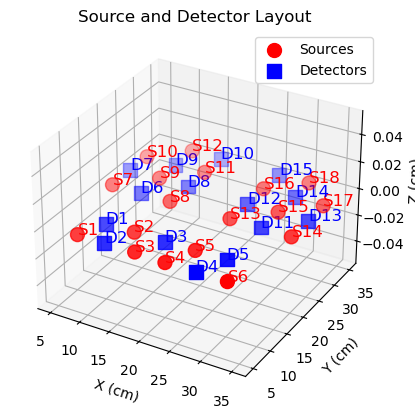
对脑电的10-20系统进行可视化,查看对应的坐标维度是否正确。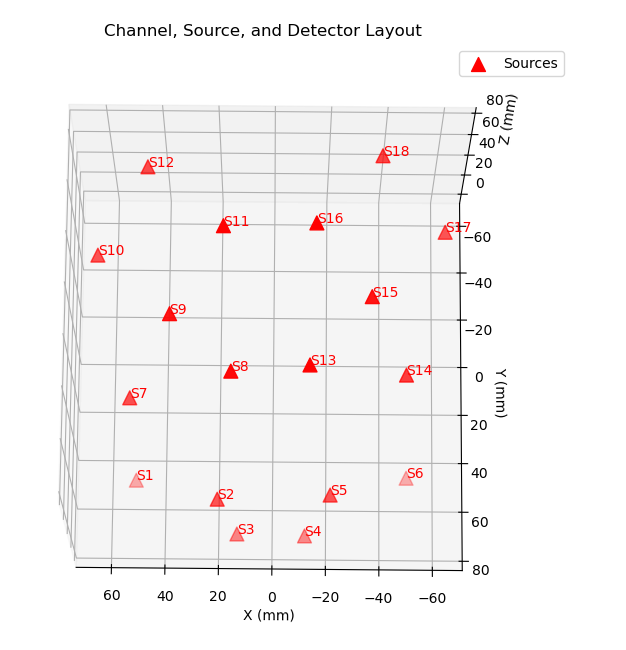
给出记录的原文件
{'CH01': [47.0, 55.0, 11.0],
'CH02': [43.0, 61.0, -1.0],
'CH03': [30.0, 61.0, 25.0],
'CH04': [28.0, 68.0, 14.0],
'CH05': [13.0, 67.0, 29.0],
'CH06': [23.0, 72.0, 4.0],
'CH07': [8.0, 71.0, 19.0],
'CH08': [-8.0, 71.0, 17.0],
'CH09': [-22.0, 71.0, 2.0],
'CH10': [-12.0, 66.0, 28.0],
'CH11': [-28.0, 66.0, 14.0],
'CH12': [-32.0, 58.0, 26.0],
'CH13': [-42.0, 59.0, -1.0],
'CH14': [-46.0, 51.0, 11.0],
'CH15': [44.0, 28.0, 48.0],
'CH16': [54.0, 16.0, 42.0],
'CH17': [24.0, 27.0, 61.0],
'CH18': [17.0, 16.0, 70.0],
'CH19': [37.0, 18.0, 61.0],
'CH20': [47.0, 5.0, 57.0],
'CH21': [25.0, 5.0, 71.0],
'CH22': [40.0, -9.0, 68.0],
'CH23': [61.0, -9.0, 48.0],
'CH24': [54.0, -24.0, 61.0],
'CH25': [17.0, -9.0, 77.0],
'CH26': [30.0, -24.0, 75.0],
'CH27': [19.0, -41.0, 79.0],
'CH28': [45.0, -39.0, 66.0],
'CH29': [33.0, -59.0, 69.0],
'CH30': [-21.0, 23.0, 65.0],
'CH31': [-14.0, 14.0, 71.0],
'CH32': [-41.0, 22.0, 55.0],
'CH33': [-51.0, 9.0, 49.0],
'CH34': [-35.0, 11.0, 64.0],
'CH35': [-24.0, 1.0, 72.0],
'CH36': [-45.0, -2.0, 60.0],
'CH37': [-38.0, -16.0, 71.0],
'CH38': [-15.0, -12.0, 78.0],
'CH39': [-27.0, -28.0, 74.0],
'CH40': [-16.0, -41.0, 79.0],
'CH41': [-59.0, -19.0, 51.0],
'CH42': [-50.0, -32.0, 62.0],
'CH43': [-40.0, -45.0, 68.0],
'CH44': [-27.0, -60.0, 70.0]}
下面是对应SD设备的
data_dir=r""
file_path = '.nirs'
file_path=os.path.join(data_dir,file_path)
data = scipy.io.loadmat(file_path)
#文件问题保存失败
#scipy.io.savemat('output_file.mat', data)
sources_data=[[51.0, 50.0, -3.0],
[20.0, 66.0, 27.0],
[13.0, 74.0, 6.0],
[-12.0, 74.0, 4.0],
[-21.0, 64.0, 27.0],
[-50.0, 48.0, -4.0],
[52.0, 29.0, 37.0],
[15.0, 27.0, 64.0],
[37.0, 5.0, 65.0],
[64.0, -24.0, 49.0],
[18.0, -25.0, 78.0],
[46.0, -56.0, 59.0],
[-13.0, 25.0, 66.0],
[-48.0, 22.0, 46.0],
[-35.0, -2.0, 66.0],
[-15.0, -26.0, 79.0],
[-62.0, -34.0, 50.0],
[-39.0, -60.0, 62.0]]
detectors_data=[[40.0, 56.0, 25.0],
[34.0, 67.0, 1.0],
[0.0, 63.0, 28.0],
[-34.0, 65.0, 1.0],
[-41.0, 50.0, 26.0],
[35.0, 28.0, 55.0],
[59.0, 3.0, 46.0],
[16.0, 4.0, 74.0],
[41.0, -23.0, 70.0],
[19.0, -58.0, 74.0],
[-33.0, 21.0, 60.0],
[-15.0, 0.0, 74.0],
[-55.0, -4.0, 52.0],
[-39.0, -30.0, 70.0],
[-14.0, -56.0, 74.0]]
data['SD']["SrcPos"][0][0]=np.array(sources_data)
data['SD']["DetPos"][0][0]=np.array(detectors_data)
#修改之后无法保存了,或者使用matlab来处理
import mne
# 创建标准 10-20 系统的电极位置
standard_montage = mne.channels.make_standard_montage('standard_1020')
# 绘制 3D 图
standard_montage.plot(kind='3d')
我们可以注意到x的分布范围是±10cm,y轴的分布范围也是±10cm。且近红外的数据分布在头部的中间的位置,所以其值域应该更小。
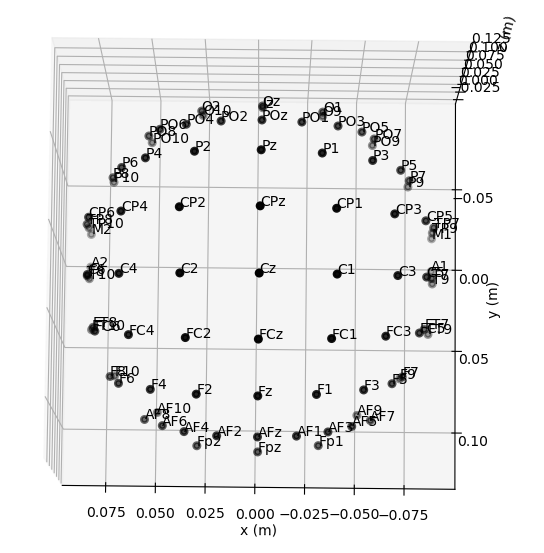
#访问某个电极的位置。
standard_montage.get_positions()["ch_pos"]["CP3"]
standard_montage.get_positions()["ch_pos"]["CP4"]
standard_montage.get_positions()["ch_pos"]["F7"]
对数据进行导入.
放弃使用python,使用matlab来导入和操作。
data = load("modified_pos_nirs1.nirs","-mat")
data.SD.SrcPos=[
51.0, 50.0, -3.0;
20.0, 66.0, 27.0;
13.0, 74.0, 6.0;
-12.0, 74.0, 4.0;
-21.0, 64.0, 27.0;
-50.0, 48.0, -4.0;
52.0, 29.0, 37.0;
15.0, 27.0, 64.0;
37.0, 5.0, 65.0;
64.0, -24.0, 49.0;
18.0, -25.0, 78.0;
46.0, -56.0, 59.0;
-13.0, 25.0, 66.0;
-48.0, 22.0, 46.0;
-35.0, -2.0, 66.0;
-15.0, -26.0, 79.0;
-62.0, -34.0, 50.0;
-39.0, -60.0, 62.0
];
data.SD.DetPos = [
40.0, 56.0, 25.0;
34.0, 67.0, 1.0;
0.0, 63.0, 28.0;
-34.0, 65.0, 1.0;
-41.0, 50.0, 26.0;
35.0, 28.0, 55.0;
59.0, 3.0, 46.0;
16.0, 4.0, 74.0;
41.0, -23.0, 70.0;
19.0, -58.0, 74.0;
-33.0, 21.0, 60.0;
-15.0, 0.0, 74.0;
-55.0, -4.0, 52.0;
-39.0, -30.0, 70.0;
-14.0, -56.0, 74.0
];
data.SD.SpatialUnit = 'mm';
new_filename = 'modified_pos_nirs1.nirs'
save(new_filename,'-struct','data')
使用sdgui可以看到被修正了。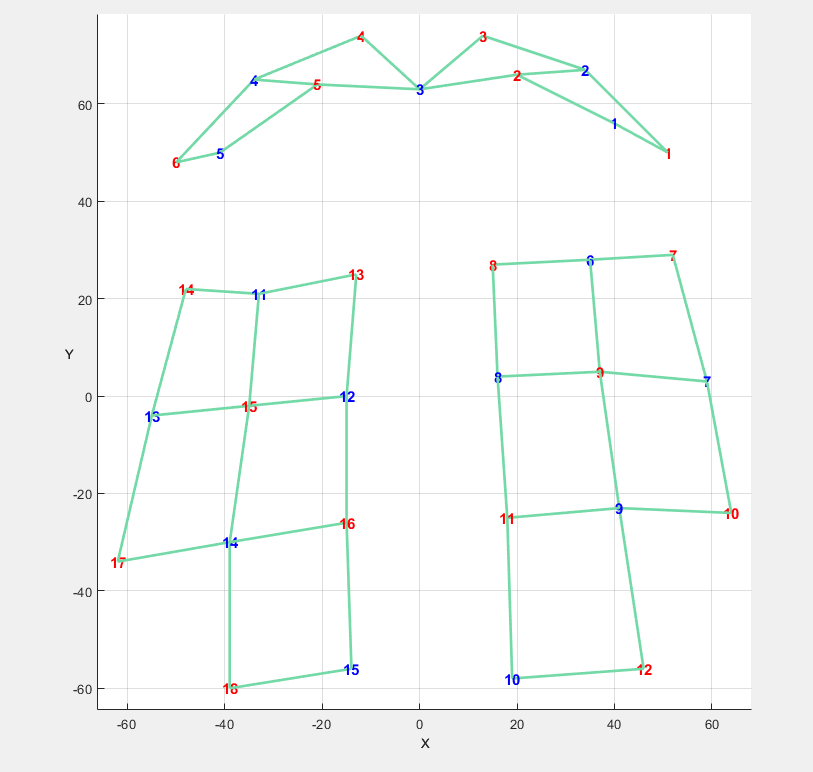
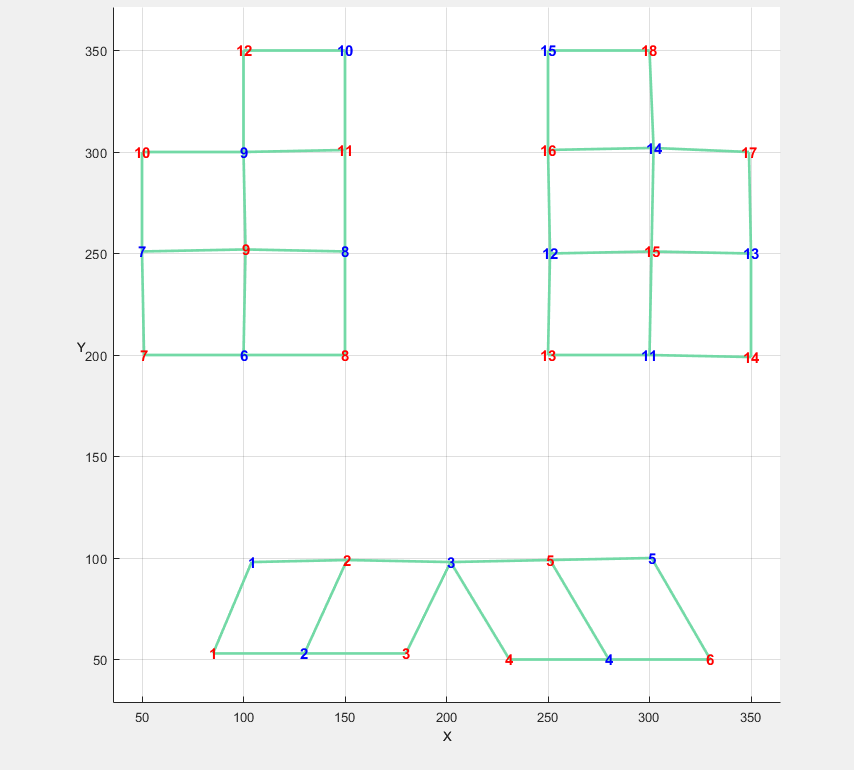
相对区别不大,但是好像镜像了,对于距离正确的影响应该不大。
预处理
需求分析,根据
procStreamGUI相关的函数
感谢您重新整理函数列表!以下是这些函数的简要介绍,按照功能分类进行说明:
1. 数据预处理
- hmrIntensityNormalized: 对光强度数据进行归一化处理,消除不同通道或时间点之间的强度差异。
- hmrIntensity2OD: 将光强度数据转换为光密度(OD)数据,这是近红外数据分析的基础步骤。
- hmrFlowInput: 处理输入数据流,通常用于数据导入和初步整理。
2. 主成分分析(PCA)
- enPCAFilter:
- 输入
dod(光密度变化数据),返回dod(滤波后数据)、svs_dod(奇异值)、nSV_dod(使用的奇异值数量)。 - 输入
dc(浓度数据),返回dc(滤波后数据)、svs(奇异值)、nSV(使用的奇异值数量)。 - 用于去除噪声或不需要的信号成分。
- 输入
3. 通道处理
- enPruneChannels: 剔除质量较差或无效的通道数据,以提高数据质量。
4. 运动伪影检测与校正
- hmrMotionArtifact: 检测运动伪影。
- hmrMotionArtifactByChannel: 按通道检测运动伪影。
- hmrMotionCorrectWavelet: 使用小波变换校正运动伪影。
- hmrMotionCorrectKurtosisWavelet: 结合峰度和小波变换校正运动伪影。
- hmrMotionCorrectPCA: 使用PCA方法校正运动伪影。
- hmrMotionCorrectPCArecurse: 递归应用PCA方法校正运动伪影。
- hmrMotionCorrectPCArecurse_Ch_dual: 在双通道数据上递归应用PCA方法校正运动伪影。
- hmrMotionCorrectPCArecurseCh_SG: 结合Savitzky-Golay滤波和PCA方法校正运动伪影。
- hmrMotionCorrectSpline: 使用样条插值校正运动伪影。
- hmrMotionCorrectSplineSG: 结合样条插值和Savitzky-Golay滤波校正运动伪影。
- hmrMotionCorrectCbsi: 使用基于通道的信号分离方法校正运动伪影。
- hmrMotionCorrectRLOESS: 使用鲁棒的局部加权回归(LOESS)校正运动伪影。
- hmrMotionCorrectSG: 使用Savitzky-Golay滤波校正运动伪影。
5. 刺激相关处理
- enStimRejection: 剔除与刺激相关的伪影或噪声。
- enStimIncData_varargin: 将刺激信息包含在数据中,用于后续分析。
6. 滤波
- hmrBandpassFilt: 应用带通滤波器去除不需要的频率成分。
- hmrBandpassFiltConc: 对浓度数据进行带通滤波。
7. 光密度与浓度转换
- hmrOD2Conc: 将光密度数据转换为血红蛋白浓度数据。
- hmrConc2OD: 将血红蛋白浓度数据转换回光密度数据。
8. 信号分析与特征提取
- enCrossCorrelation: 计算通道之间的互相关性,用于分析信号之间的关系。
- enAdaptiveFilteringSS: 使用自适应滤波方法去除噪声。
- hmrCreateAuxRegressor: 创建辅助回归器,用于回归分析。
- hmrBlockAvg: 对数据进行块平均,通常用于提高信噪比。
- plotTrials: 绘制试次数据,用于可视化分析。
- hmrFindHrfOutlier: 检测和剔除与血流动力学响应函数(HRF)相关的异常值。
9. 回归与反卷积
- hmrRegressors: 创建回归器,用于回归分析。
- hmrDeconvTB_3rd: 使用三阶泰勒展开进行反卷积。
- hmrDeconvTB_SS3rd: 结合三阶泰勒展开和稳态方法进行反卷积。
- hmrDeconvTB_SS3rd_Highest: 结合三阶泰勒展开、稳态方法和最高响应进行反卷积。
- hmrDeconv_SS: 使用稳态方法进行反卷积。
- hmrDeconvHRF_Drift: 考虑漂移的HRF反卷积。
- hmrDeconvHRF_DriftSS: 结合漂移和稳态方法的HRF反卷积。
10. 图像重建与可视化
- hmrImageHrfMeanTwin: 计算HRF的平均时间窗口图像。
- hmrImageRecon: 进行图像重建,通常用于空间分析。
总结
这些函数涵盖了从数据预处理、运动伪影校正、信号分析到回归与反卷积的完整流程。根据具体的研究需求,可以选择合适的函数组合来处理和分析近红外数据。例如:
- 如果需要去除运动伪影,可以使用
hmrMotionCorrectWavelet或hmrMotionCorrectPCA。 - 如果需要分析血红蛋白浓度变化,可以使用
hmrOD2Conc和hmrBandpassFiltConc。 - 如果需要反卷积分析,可以使用
hmrDeconvTB_SS3rd或hmrDeconvHRF_DriftSS。
下个章节在进行记录。

魔乐社区(Modelers.cn) 是一个中立、公益的人工智能社区,提供人工智能工具、模型、数据的托管、展示与应用协同服务,为人工智能开发及爱好者搭建开放的学习交流平台。社区通过理事会方式运作,由全产业链共同建设、共同运营、共同享有,推动国产AI生态繁荣发展。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容







所有评论(0)